2018-08-01
阅读数:18780
为抵御各种各样的病原体,人类或哺乳动物的适应性免疫系统拥有大量的T细胞受体(TCRs)和B细胞受体(BCRs),统称为免疫组库。高通量测序(NGS)技术允许对T或B细胞克隆进行深入分析,能够在检测T或B细胞时提供前所未有的精细度。

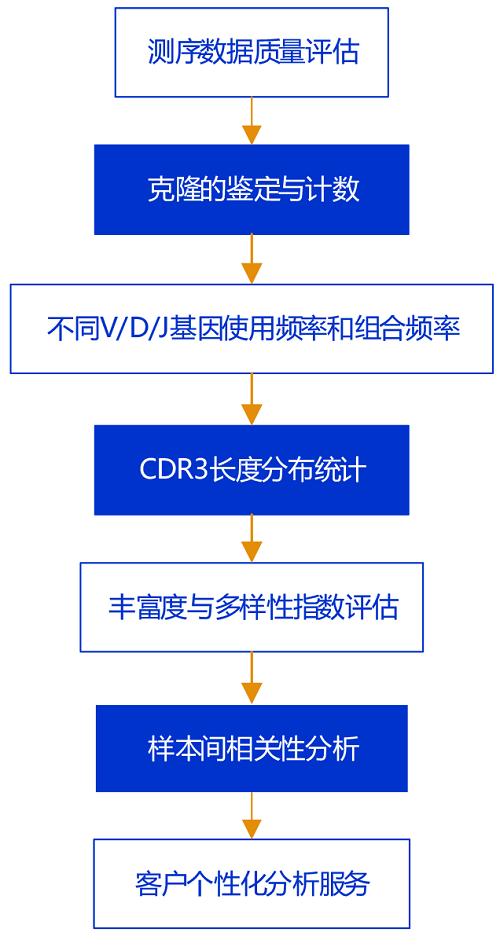
该技术可以有效评估TCR或BCR互补决定区域(CDR3)的多样性,并评估克隆类型的构成:包括特定克隆类型的比例;克隆之间的相似程度;V(D)J基因的使用频率;核苷酸插入与缺失;CDR3区域长度分布等信息。因此,免疫组库高通量测序为评估健康或疾病状态下的机体适应性免疫系统提供了可能。
高通量测序技术可快速便捷地对免疫组库进行全景式的扫描,但其涉及的PCR反应会导致假阳性增加以及扩增偏倚,从而对数据结果的准确性造成潜在影响。
目前,现有的免疫组库技术主要分为两类:
1.基于多重PCR的文库构建方案:以DNA为模板,针对可变区(CDR3)设计多对引物,扩增出CDR3区段,再进行文库构建并测序;
2.基于5'RACE的文库构建方案:以mRNA为模板,在恒定区设计引物逆转录扩出TCR或BCR的全长序列,经过两轮PCR富集这一目的序列,再进行文库构建并测序。
但是,现有的基于多重PCR的文库构建方案存在明显不足之处:该方法同时进行几十种引物的扩增,体系异常复杂;多重引物设计需已知参考序列,不能充分捕捉到人类等位基因突变序列;多种引物扩增效率可能存在偏倚,难以等效扩增;与使用mRNA构建文库相比,DNA所需的PCR反应体系较大,样本要求较高。
技术升级一:
广州华银健康科技有限公司推出全新的免疫组库测序技术服务,基于5'RACE原理,并在原始模板扩增前引入UMI(Unique Molecular Indices,分子条形码)进行文库构建:以恒定区单引物逆转录扩增出CDR区
域全长序列的同时,引入UMI序列,可对后续PCR反应或测序过程中引入的错误进行有效纠正,从而提高准确率。
技术升级二:
在逆转录环节,广州华银健康科技有限公司设计了针对微量样品(低至100个细胞)的实验流程:即通过oligo dT磁珠捕获mRNA,以磁珠上的oligo dT为逆转录引物合成cDNA,并纯化回收cDNA,较常规方法可大大提高效率,保证了人或小鼠来源的微量样品的实验成功率。
全新免疫组库测序技术服务亮点
1.可满足微量样品建库的实验要求(最低可至100个细胞),兼容人以及小鼠样本
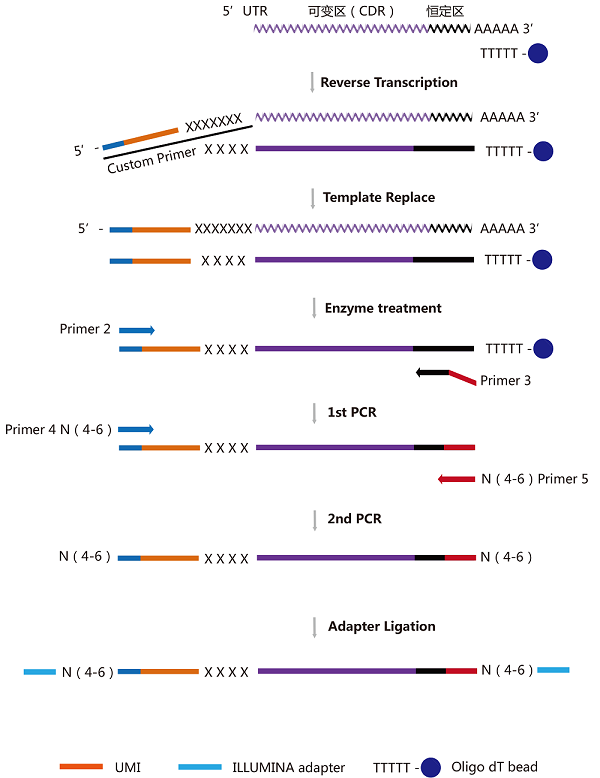
微量RNA起始建库流程图
2.低RNA起始量的样本也能快速达到测序饱和期,相同RNA起始量下的样本其相关度较高
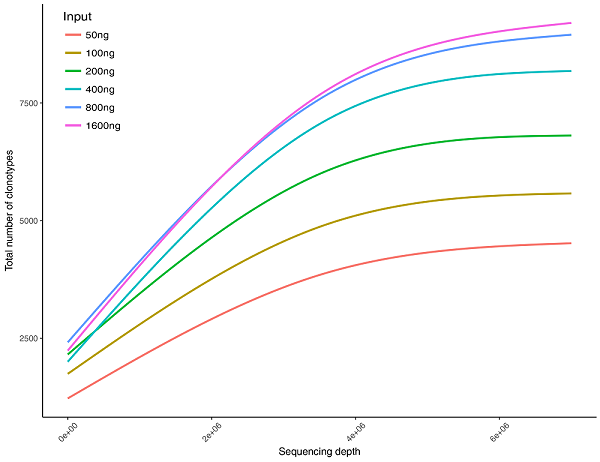
同一样本RNA起始量不同时到达测序饱和期数据统计曲线图
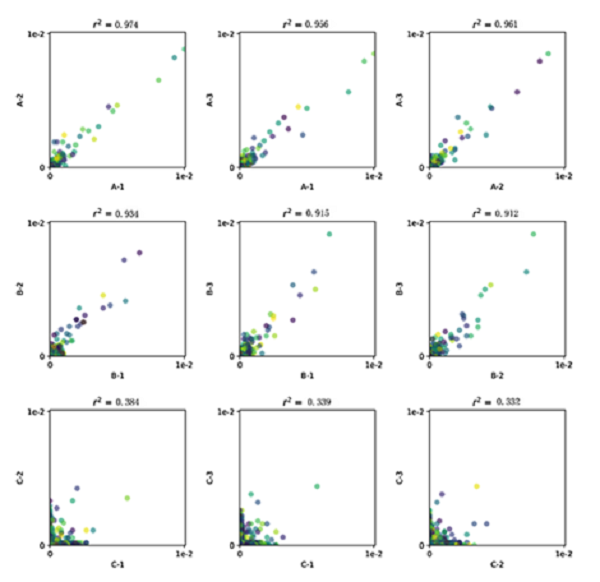
同一样本RNA起始量相同时样本间的相关性图
(A1/A2/A3为起始RNA量为1600ng; B1/B2/B3为起始量为400ng; C1/C2/C3为起始量为50ng)
3.基于强大的UMI标记与校正算法,可有效纠正PCR与测序过程引入的错误与偏倚,极大地提高准确度
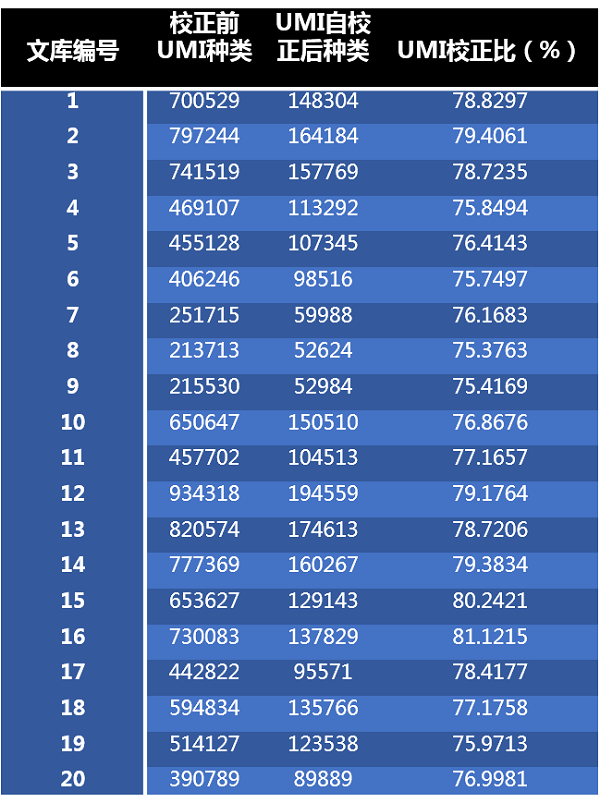
经过UMI算法校正后的部分测试数据统计表
4.对测序下机数据进行多维度分析,给出最全面的数据结果报告,并可定制个性化分析服务
参考文献:
1.Abbas A K, et al. Cellular & Molecular Immunology 6e[J].
2.Hou X L, et al. Current status and recent advances of next generation sequencing techniques in immunological repertoire[J]. Genes & Immunity, 2016, 17(3):153.
3.Lefranc, et al. "IMGT?, the international ImMunoGeneTics information system?." Nucleic acids research 37.suppl_1 (2008): D1006-D1012.
4.Chunlin Wang, et al. High throughput sequencing reveals a complex pattern of dynamic interrelationships among human T cell subsets. PNAS, 2010, vol. 107:1518–1523.
5.Jennifer Benichou, et al. Rep-Seq: uncovering the immunological repertoire through next-generation sequencing. Immunology, 2011, 135: 183–191.
6.Jeroen W J van Heijst, et al. Quantitative assessment of T cell repertoire recovery after hematopoietic stem cell transplantation. Nature Medicine, 2013,VOL.19: 372-379.
7.Miran Jang, et al. Characterization of T cell repertoire of blood,tumor, and ascites in ovarian cancer patients using next generation sequencing. OncoImmunology, 2015, Vol.4:e1030561-1-10.
8.Evaggelia Liaskou, et al. High-throughput T-cell receptor sequencing across chronic liver diseases reveals distinct disease-associated repertoires. Hepatology, 2015,1-10.
9.Bates S T, et al. Global biogeography of highly diverse protistan communities in soil[J]. The ISME journal, 2013,7(3): 652-659.






