
技术介绍
染色质转座酶可及性测序(Assay for Transposase-Accessible Chromatin with high throughput sequencing, ATAC-seq)是一种创新的表观遗传学研究技术,由美国Stanford大学的William Greenleaf教授在2013年所研发,该技术利用DNA转座酶Tn5酶对某种特定时空下开放的核染色质区域进行切割,并结合高通量测序技术来研究染色体的开放性,进而获得在该特定时空下基因组中所有活跃转录的调控序列。
结合10X Genomics测序平台,Chromium单细胞核ATAC测序技术(snATAC-seq)可以帮助研究者在单细胞水平上分析染色质开放性,逐个细胞地揭示表观基因组景观,从而获得细胞类型和状态的信息并深入探究基因调控机制。snATAC-seq可广泛应用于癌症、免疫、干细胞、胚胎分化发育和神经分化发育等表观遗传修饰的研究,为疾病或遗传发育等研究提供了新的思路。
研究方向
? 定义细胞类型和细胞状态
? 鉴定主要的调控因子
? 探索顺式调控元件,如启动子和增强子
? 描绘基因调控网络
技术优势
? 单细胞分辨率
跨越群体的平均数信息,获得单个细胞核的表观基因组图谱
? 高捕获效率
每个样本细胞核捕获率可达60%以上
? 兼容多类型样本
经验证适用于细胞系、原代细胞、新鲜或冷冻组织的组织样本
? 低成本
相较于其他单细胞核捕获平台或传统酶切方法,10X平台成本更低
项目流程
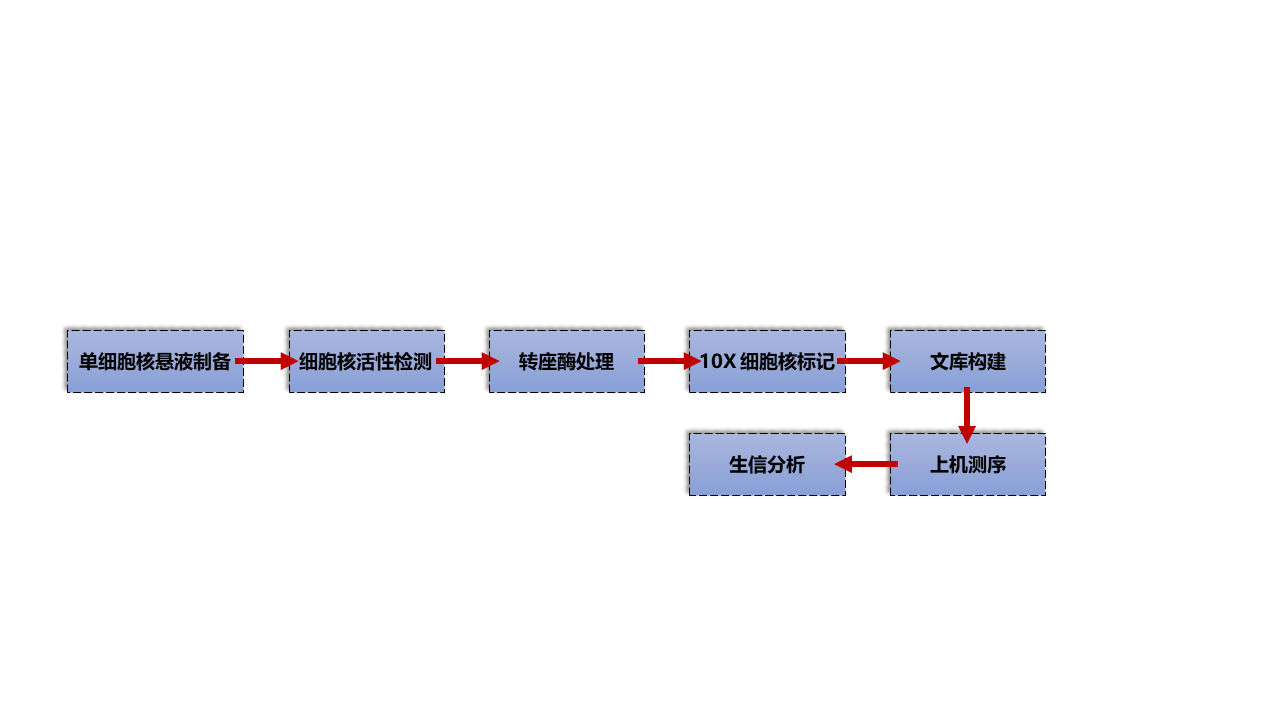
送样建议
样本来源:人、大鼠和小鼠(其他样本类型请咨询)
新鲜或冻存的组织样本:黄豆大小;
单细胞悬浮液样本:500-2,000个细胞/μL、细胞小于40μm、细胞数大于106、细胞活性大于80%;
单细胞细胞核悬浮液样本:3,000-5,000个细胞核/μL、体积大于50μL、核膜完整性较好;
结果展示
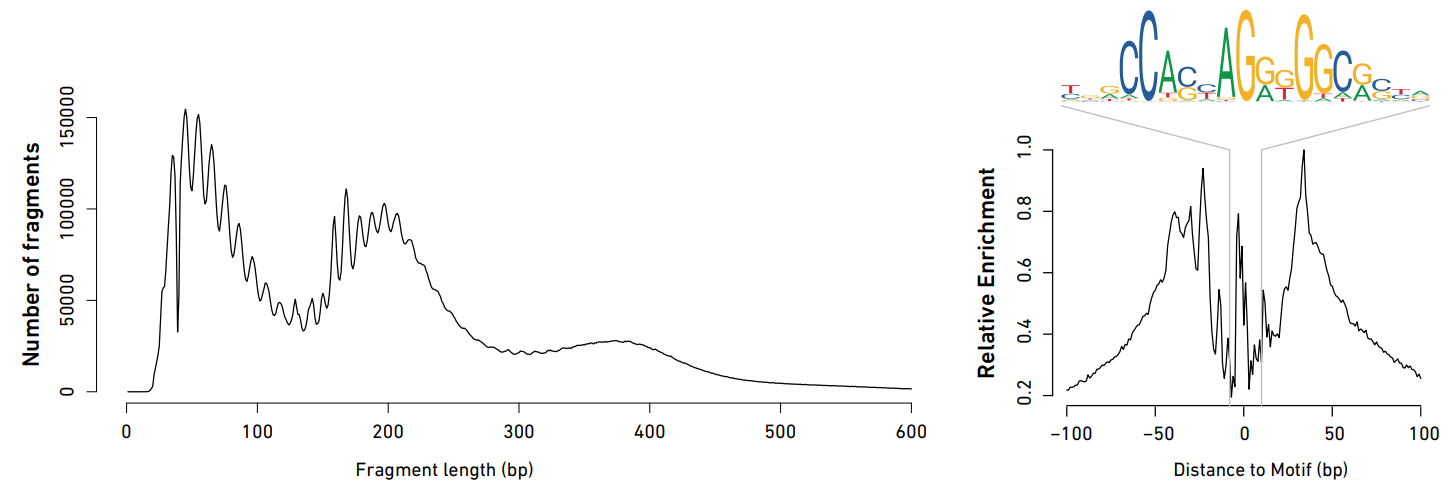
图1 高质量的单细胞ATAC-seq文库。左图展示了GM12878细胞系的单细胞ATAC-seq文库的插入片段大小分布情况,峰的出现以每184个碱基对的长度作为周期,揭示了单个核小体和核小体多聚体对基因组的保护情况;右图展示了GM12878细胞系中预测的转录因子 (CTCF) 结合位点的足迹信号的累积情况。
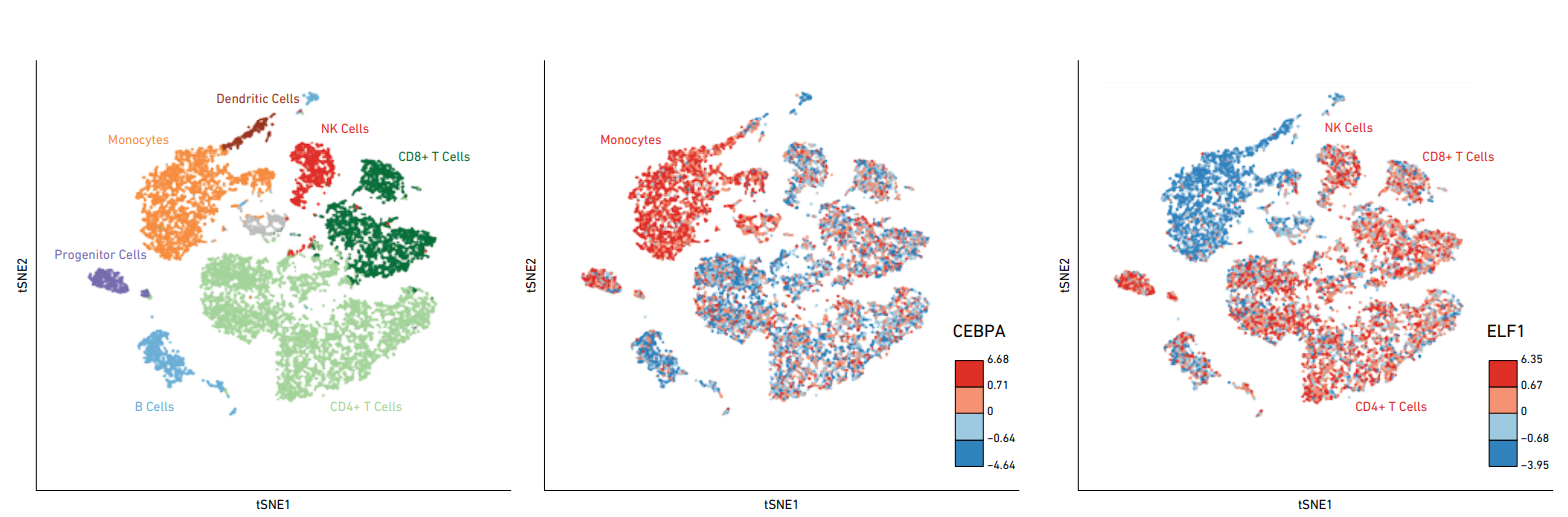
图2 外周血单个核细胞 (PBMC) 的染色质开放性图谱。左图来自健康捐赠者的约10,000个 PBMC的t-SNE聚类结果。基于充分表征的 TF 基序和细胞类型特异性峰的富集分数确定主要亚群;中图和右图展示了表面覆盖有CEBPA(单核细胞标志物)和 ELF1(自然杀伤T 细胞标志物)的PBMC的染色质开放性图谱,其中PBMC由20%的单核细胞和66%的自然杀伤T细胞组成。
经典案例

英文标题:Developmental and evolutionary dynamics of cis-regulatory elements in mouse cerebellar cells 1
发表期刊:Science (IF=47.73)
在传统认识中,小脑主要起着维持身体平衡、和协调躯体运动的作用,除此之外,小脑还参与其他复杂动作的过程,如语言和记忆等。小脑在发育过程中,生发区以时空受限的方式产生不同的神经元和神经胶质细胞群——脑室区和菱形唇。细胞类型的特化和分化过程由增强子和启动子等顺式作用元件 (CRE)、以及与它们结合的转录因子 (TF) 所调控。单细胞方法已被用于研究大脑的基因调控机制,包括发育中的小鼠以及成年小鼠大脑和人类大脑,但一直缺乏对哺乳动物器官发育过程的全面描述。
在本项研究中,作者首先使用了snATAC-seq技术来描绘小鼠出生前后、跨越11个阶段的小脑发育时期中约90,000个细胞的染色质可及性的景观(代表CRE的活性),该CRE活性特征图谱突出了基因调控过程中在细胞水平和时间维度上的特异性,发现了细胞分化的命运与小脑祖细胞的时空异质性有关。然后,作者将上述snATAC-seq数据关联脊椎动物基因组进行进化分析,发现CRE序列在小脑发育过程中保守性下降,这可能是由于小脑细胞分化导致的。这项研究评估了小鼠小脑基因调控下的发育和进化动力学,并为哺乳动物器官发育提供了新见解。
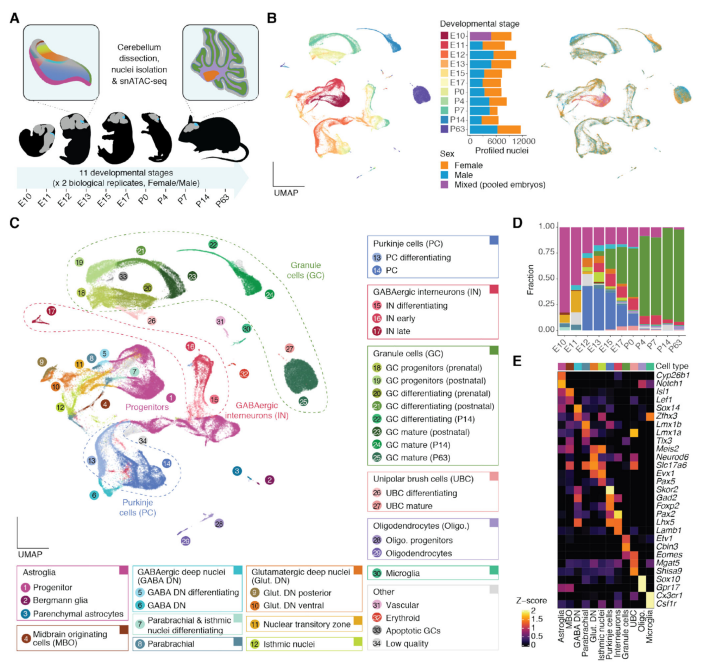
图3 小鼠小脑发育的snATAC-seq图谱
1.Sarropoulos I, Sepp M, Fr?mel R, et al. Developmental and evolutionary dynamics of cis-regulatory elements in mouse cerebellar cells. Science (1979). 2021;373(6558). doi:10.1126/science.abg4696